Neuer Ansatz zur Strukturbestimmung von Makromolekülen mit höherer Auflösung in ihrem nativen Kontext
Eine optimierte Auflösung im Subnanometerbereich durch Hybrid-Subtomogramm-Mittelung - Einzelpartikel-Kryo-Elektronenmikroskopie
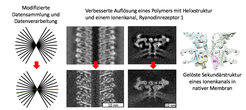
Die Kryo-Elektronentomographie wurde mit der Subtomogramm-Mittelung (StA) kombiniert und führte im nativen Kontext zu hochaufgelösten Strukturen von Makromolekülen. Hochauflösende StA ist jedoch aufgrund der strahleninduzierten Probendrift, Bildern mit schlechten Signal-Rausch-Verhältnissen (SNR), Herausforderungen bei der Korrektur der Kontrastübertragungsfunktion (CTF) und einer begrenzten Partikelanzahl schwer zu erhalten. In dieser Arbeit wurden diese Probleme angegangen, indem Neigungsreihen mit einer höheren Elektronendosis bei der Null-Grad-Neigung gesammelt wurden. Die Partikel wurden dann in rekonstruierten Tomogrammen lokalisiert, mit konventioneller StA verarbeitet und aus den hochdosierten Bildern in 2D wieder extrahiert. Anschließend wurden Einzelpartikel-Analyseverfahren angewendet, um die 2D-Partikelausrichtung zu verfeinern und eine Rekonstruktion zu erhalten. Der Einsatz dieser Hybrid-Subtomogramm-Mittelung (hStA) verbesserte die Auflösung zum Beispiel von der Struktur des Tabakmosaikvirus von 7,2 auf 4,4 Å und für den Ionenkanal Ryanodinrezeptor 1, RyR1 in dicht gepackten nativen Membranen von 12,9 auf 9,1 Å. Diese höhere Auflösung macht hStA zu einer vielversprechenden Methode für weitere StA-Projekte, die auf eine Auflösung im Subnanometerbereich abzielen.
Die Elektronentomographie von biologischen Proben, die in ihrer hydratisierten Form vorliegen, ermöglicht die Beobachtung von Molekülen in ihren nativen Zuständen wie zum Beispiel von intrazellulären Proteinen, Membranproteinen und Desoxyribonukleinsäure (DNA). Moleküle, die in mehreren Kopien von tomographischen Volumen vorhanden sind, können in einem als "Subtomogramm-Mittelwertbildung" bezeichneten Prozess aufeinander abgestimmt und gemittelt werden, um ihre Struktur zu analysieren und die Mechanismen ihrer Funktionsabläufe zu verstehen. Es müssen jedoch einige Anforderungen bewältigt werden, um informative hochaufgelöste Strukturen zu erhalten: optische und geometrische Verzerrungen, aufwendige Bildverarbeitung und begrenzter Durchsatz bei der Datenerfassung gehören zu den dabei auftretenden Problemen. Andererseits erfasst die bisher gut etablierte Methode "Einzelpartikel-Kryo-Elektronenmikroskopie (Kryo-EM)" gereinigte Proteine, die in einer Schicht aus amorphem Eis in 2D eingefroren sind, und führt eine dreidimensionale (3D) Rekonstruktion aus mehreren Proteinkopien durch, die aus verschiedenen Richtungen betrachtet werden. Kryo-EM erhielt breite Anerkennung und die Entwickler wurden 2017 mit dem Nobelpreis für Chemie ausgezeichnet. Kryo-EM führt zu hochaufgelösten Strukturen, basierend auf Makromolekülen, die aus ihrem nativen Kontext extrahiert wurden und zu nicht-nativen Konformationen führen kann.
Forschende des Max-Planck-Instituts für Biophysik in Frankfurt am Main verbesserten die Arbeitstechniken für Strukturbestimmungen aus Kryo-Elektronentomogrammen, indem sie Vorteile der Mittelung von Subtomogrammen mit der Einzelpartikel-Kryo-Elektronenmikroskopie kombinierten. Für jedes 2D-Bild eines Einzelpartikels folgt die Aufnahme eines Tomogramms. Die betreffenden 3D-Tomogramme der Makromoleküle werden gegenseitig aufeinander ausgerichtet. Die letzten Verfeinerungsschritte werden jedoch unter Verwendung entsprechender Software aus den Einzelpartikel-ähnlichen Bildern durchgeführt. Dieser Arbeitsablauf ermöglicht eine Driftkompensierung, die während der Elektronenstrahl-Exposition am amorphem Eis auftritt als auch bessere Rekonstruktionen, was schließlich zu einer höheren Auflösung führt. Diese Hybridtechnik beschleunigt auch die Datenerfassung und ermöglicht einen höheren Durchsatz bei den sehr teuren modernen Kryo-Elektronenmikroskopen.
„Unser Ziel ist es, Rekonstruktionen im Subnanometerbereich zu erhalten, da wir mit dieser verbesserten Auflösung zum Beispiel auch alpha-Helices von medizinisch relevanten Membranproteinen definieren können, die Kanäle für Natrium-, Kalium- oder Kalziumionen bilden. Wir konnten die Auflösung verbessern, indem wir unsere Datenerfassungs- und -verarbeitungsroutinen modifiziert haben, und jetzt freuen wir uns, unsere Erfahrungen an die breitere Strukturbiologie-Gemeinschaft weiterzugeben “, sagt der Leiter der Forschungsgruppe, Misha Kudryashev. „Wir glauben, dass diese Methode in Zukunft von uns und den Kollegen genutzt wird, um einzigartige Einblicke in die Struktur und Funktion von Makromolekülen in ihrem ursprünglichen Kontext zu gewinnen.“
