Flexibel und gut abgeschirmt - Neueste Forschungsergebnisse zum SARS-CoV-2-Protein werfen unerwartetes Licht auf die Infektionsstrategie des Virus
Neue Erkenntnisse zeigen, wie das Virus Zellen infizieren kann
Im Kampf gegen das Coronavirus SARS-CoV-2 haben sich Forscher aus verschiedenen Forschungseinrichtungen in Deutschland zusammengeschlossen, um das Spike-Protein auf der Oberfläche des Virus zu untersuchen - dieses Protein ermöglicht es dem Virus, an menschliche Zellen anzudocken und diese zu infizieren. Dabei haben sie überraschende Erkenntnisse gewonnen, darunter eine nicht vermutete Biegsamkeit des Spike-Proteins auf der Zelloberfläche, sowie eine effektive physikalische Abschirmung, die die Erkennung der Infektion durch das Immunsystem erschwert. Über die Ergebnisse berichtet Science in seiner Onlineausgabe vom 18.08.2020.
Um an menschliche Zellen zu binden und diese zu infizieren, benötigt das Coronavirus SARS-CoV-2 ein spezielles Protein auf seiner Oberfläche. Diese stachelförmigen Strukturen („Spikes“) stehen im Mittelpunkt der Impfstoffentwicklung, da durch sie eine Immunantwort beim Menschen induziert wird. Aus diesem Grund haben Wissenschaftler des Max-Planck-Instituts für Biophysik, des EMBL Heidelberg und des Paul-Ehrlich-Instituts ihre Forschungsaktivitäten insbesondere auf die Oberflächenstruktur des Virus konzentriert, um Erkenntnisse sowohl für Impfstoffe als auch für die Entwicklung wirksamer Therapeutika zur Behandlung infizierter Patientinnen und Patienten zu gewinnen. Die Bestimmung der räumlichen Strukturen ist Grundlage für das bessere Verständnis der einzelnen Schritte des Infektionsprozesses, wie beispielsweise das Eindringen in die menschliche Wirtszelle.
Die Kombination von Cryo-Elektronentomographie, Subtomogramm-Mittelung und Molekularen Simulationen ermöglichte die Analyse der Struktur der Spike-Proteine in ihrer natürlichen Umgebung und mit nahezu atomarer Auflösung. Hierzu wurden 266 Cryo-Tomogramme von etwa 1000 verschiedenen Viren erzeugt, die jeweils im Durchschnitt 40 Spike-Proteine auf ihrer Oberfläche trugen. Die Mittelwertbildung der Subtomogramme und der Bildbearbeitung lieferten schließlich die benötigten wichtigen Strukturinformationen über die Spikes.

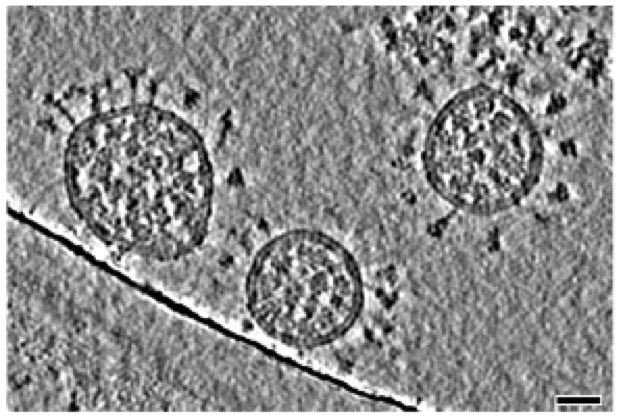
Schnitt durch ein Kryo-Elektronentomogramm von SARS-CoV-2-Partikeln. Adaptiert von Turoňová et al. Maßstabsleiste 30 nm.
Ein überraschendes Ergebnis: Die Daten zeigten, dass der kugelförmige Teil des Spike-Proteins, der die rezeptorbindende Region und die für die Fusion mit der Wirtszelle notwendigen Komponenten enthält, über einen flexiblen Stiel mit der Virusoberfläche verbunden ist. "Der obere kugelförmige Teil des Spike-Proteins hat eine Struktur, die von rekombinanten Proteinen, die für die Impfstoffentwicklung verwendet werden, gut reproduziert wird", erklärt Martin Beck, Gruppenleiter am EMBL und Direktor am MPI für Biophysik. "Neu waren jedoch die Erkenntnisse über den Stiel, der den kugelförmigen Teil des Spike-Proteins an der Virusoberfläche fixiert".
"Wir hatten erwartet, dass der Stiel starr ist", ergänzt Gerhard Hummer, Direktor am MPI für Biophysik und Professor am Institut für Biophysik der Goethe-Universität Frankfurt. "In unseren Computermodellen und Elektronentomogrammen haben wir aber entdeckt, dass der Stiel extrem flexibel ist. Die Spike-Proteine stehen nicht senkrecht auf der Membran, sondern sind in alle Richtungen geneigt." Das Team identifizierte vier verschiedene Domänen (Abschnitte) im Stiel, die sie als Hüft-, Knie-, Knöchel-Domäne, und schließlich als in die Membran eingebettete Fuß-Domäne bezeichneten. Durch die Kombination von Molekularen Simulationen und Cryo-Tomographie wiesen sie nach, dass diese Domänen Biegebewegungen ausführen können, was die Flexibilität der Spikes erklärt.
Vier Spike-Proteine auf der Membranoberfläche von Sars-CoV-2.
Atomistische Molekulardynamik-Simulationstrajektorie von vier in eine Membran eingebetteten Spike-Proteinen. Die Proteine und Lipide werden in Oberflächendarstellung gezeigt. Glykane sind durch grüne Van-der-Waals-Perlen dargestellt. Wasser und Ionen wurden der Übersichtlichkeit halber weggelassen. Darstellung in 600ns Simulationszeit. Sören von Bülow, Mateusz Sikora, Gerhard Hummer, MPI für Biophysik.
"Wie ein Ballon an einer Schnur scheinen sich die Spike Proteine auf der Oberfläche des Virus zu bewegen und können so nach dem Rezeptor für das Andocken an die Zielzelle suchen", beschreibt Jacomine Krijnse Locker, Gruppenleiterin am Paul-Ehrlich-Institut, die Ergebnisse. Die Analysen zeigten schließlich, dass der Stiel mit vielen Glykan-Ketten ausgestattet ist. Diese könnte dem Stiel zudem eine Art schützenden Mantel aus Kohlenhydraten verleihen und ihn so vor neutralisierenden Antikörpern verstecken. Dies ist eine weitere wichtige Erkenntnis auf dem Weg zu wirksamen Impfstoffen und Medikamenten.
Quelle:
Beata Turoňová et al. In situ structural analysis of SARS-CoV-2 spike reveals flexibility mediated by three hinges Science, published on 18 August 2020
DOI: 10.1126/science.abd5223