Elektronen-Cryomikroskopie von Membranproteinkomplexen
Forschungsbericht (importiert) 2019 - Max-Planck-Institut für Biophysik
Elektronen-Cryomikroskopie (CryoEM) ist die Methode der Wahl zur Bestimmung der Struktur von Membranproteinkomplexen, die für die Röntgenkristallographie zu instabil oder zu dynamisch sind. Intakte ATP-Synthasen beispielsweise konnten bislang einer Kristallisation widerstehen, dank CryoEM sind aber nun zentrale Aspekte ihrer Mechanismen deutlich geworden. Die beiden am besten aufgelösten und damit informativsten Strukturen, und zwar die der Chloroplasten ATP-Synthase und eines Dimers der mitochondrialen ATP-Synthase, kommen aus unserer Abteilung.
CryoEM von Membranproteinkomplexen
Alle lebenden Zellen und ihre Organellen sind von Lipidmembranen umgeben. Membranproteine und ihre Komplexe, die in die 4 nm-Lipid-Doppelschicht eingebettet sind, sind an praktisch allen biochemischen und biophysikalischen Prozessen in der Zelle maßgeblich beteiligt. Dazu gehören Energieumwandlung, Stoffaustausch, Zellfusion und -teilung, sensorische Wahrnehmung, Signaltransduktion oder neuronale Prozesse, um nur einige zu nennen. Ein genaues Verständnis jedes dieser Prozesse erfordert die genaue Kenntnis der Struktur der beteiligten Proteine, vorzugsweise in verschiedenen Funktionszuständen. Das Hauptziel unserer Abteilung Strukturbiologie ist es, die Struktur und die molekularen Mechanismen von Membranproteinen und großen Membranproteinkomplexen zu verstehen. Unsere Hauptwerkzeuge sind die CryoEM und die Membranbiochemie.
In den letzten fünf Jahren hat die CryoEM eine Revolution erfahren (The Resolution Revolution; [1]), die durch die Entwicklung von Direktelektronendetektoren und neuer Bildverarbeitungssoftware ausgelöst wurde. Unsere Abteilung war von Anfang an direkt an dieser atemberaubenden Entwicklung beteiligt.
CryoEM-Struktur der Chloroplasten ATP-Synthase
Die Chloroplasten grüner Pflanzen wandeln Sonnenenergie in chemische Energie um. Photosynthetisch generiertes Adenosintriphosphat (ATP) ist die primäre Quelle biologisch nutzbarer Energie auf der Erde. Pflanzen produzieren ATP mithilfe der F1Fo ATP-Synthase der Chloroplasten (cF1Fo). Der 500 kDa große Komplex aus 26 Untereinheiten wird durch den elektrochemischen Protonengradienten über die Thylakoidmembran und die ATP-Synthese im hydrophilen α3β3-Kopf (cF1) durch den cFo-Drehmotor in der Membran angetrieben. cFo enthält einen Rotorring aus 14 c-Untereinheiten mit jeweils einem konservierten protonierbaren Glutamat. Die Untereinheit a leitet die Protonen zu und von den Protonierungsstellen des c-Rings. Der zentrale Schaft aus den Untereinheiten γ und ε überträgt das Drehmoment vom Fo-Motor auf den katalytischen cF1-Kopf, was zur Synthese von drei ATP pro Umdrehung führt [2].
Wir bestimmten die CryoEM-Struktur des intakten cF1Fo-Komplexes aus Spinat in Lipid Nanodiscs mit einer Auflösung von 2,9 Å (cF1) bis 3,4 Å (cFo) und bauten ein vollständiges atomares Modell ([3]; Abb. 1 A, B). In cF1 waren die gebundenen Nukleotide, Mg2+-Ionen und Wassermoleküle aufgelöst (Abb. 1 C, D). Die Untereinheit δ verbindet die drei α-Untereinheiten mit dem peripheren Stator. Der Zugangskanal zu den protonierten Glutamatresten des c-Rings führt durch eine lange, membran-intrinsische Haarnadel aus zwei nahezu horizontalen α-Helices der Untereinheit a, die wir in der mitochondrialen ATP-Synthase von Polytomella entdeckt hatten [4]. Wie wir heute wissen, ist diese Haarnadel ein Merkmal aller Rotations-ATPasen [2].

Abb. 1: CryoEM-Struktur der Chloroplasten F1Fo ATP-Synthase. A, B: Zwei zueinander senkrechte Ansichten der 2,9 - 3,4 Å-Karte des monomeren Komplexes mit dem c-Ring (gelb) in der Membran (graues Band). Der F1-Kopf (grün) ist mit der Untereinheit a (hellblau) durch den flexiblen peripheren Schaft (violett) verbunden, der aus zwei langen, verschlungenen Helices besteht, die über die Untereinheit δ (orange) mit dem F1-Kopf verbunden sind. Der aus den Untereinheiten γ (blau) und ε (magenta) bestehende zentrale Stiel überträgt das Drehmoment vom c-Ring-Rotor auf F1. C, D: Gebundenes Adenosindiphosphat (ADP) in den katalytischen Bindungstaschen der Untereinheiten β mit gelösten Mg-Ionen und Wassermolekülen. ATP wurde während der Isolierung zu ADP hydrolysiert [3].
Struktur und Funktion der dimeren mitochondrialen ATP-Synthase aus Polytomella sp.
Die größte und stabilste bekannte ATP-Synthase stammt aus Mitochondrien der einzelligen Alge Polytomella sp. Der Komplex eignet sich ideal für die hochauflösende CryoEM. Unsere aktuelle CryoEM-Karte des Polytomella-Dimers (Abb. 2 A) bietet mit 2,7 Å die bisher höchste Auflösung für eine vollständige, funktionsfähige ATP-Synthase [5]. Die Struktur klärt eine Reihe bisher ungelöster Fragen zur Energieumwandlung in ATP-Synthasen. Eine Schlüsselfrage war beispielsweise, wie bei der stöchiometrischen Diskrepanz zwischen dem c-Ring in Fo (bestehend aus 8 bis 17 c-Untereinheiten) und dem dreifach symmetrischen F1-Kopf bei der Synthese von ATP, die auf Rotation beruht, effizient und elastisch gekoppelt wird. Weitere Fragen bezogen sich auf den Weg, auf dem Protonen durch die Membran gelangen sowie auf den Mechanismus, der den Fo-Rotor antreibt [2].
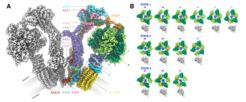
Abb. 2: CryoEM-Struktur des Dimers der mitochondrialen ATP-Synthase aus Polytomella. A: Seitenansicht der 2,7 Å-Elektronendichte-Karte mit farbig dargestellten Untereinheiten auf der rechten Seite. Das Dimer biegt die Lipid-Doppelschicht (graues Band). Der massive Stator besteht aus zehn einzigartigen ASA-Untereinheiten (ATP synthase-associated proteins), darunter die von uns neu entdeckte Komponente ASA10 (rot), die das Dimer in der Membran auf der luminalen Seite zusammenhält. B: Die dreizehn Unterzustände der primären Rotationszustände 1, 2 und 3, die durch Bildverarbeitung und 3D-Klassifikation aufgelöst werden konnten. Untereinheit β, hellgrün; α, dunkelgrün; γ, blau; Stator grau [5].
Ein vollständiges atomares Modell des 1,6 MDa großen Dimers zeigt 62 Untereinheiten, viele davon erstmals, da es bislang keine bekannten Strukturen von Homologen der 10 peripheren Stator-Untereinheiten gab (Abb. 2 A). Es gelang uns, 13 unabhängige Rotationszustände zu trennen (Abb. 2 B), die eine detaillierte molekulare Beschreibung der Rotationsbewegungen ermöglichen. Völlig unerwartet stellten wir dabei fest, dass sich der F1-Kopf zusammen mit dem zentralen Schaft und dem c-Ring zu Beginn jedes der drei 120°-Schritte um etwa 30° dreht. Die flexible Kopplung des F1-Kopfes an den Fo-Motor wird durch das neu entdeckte Scharnier im OSCP-Protein (oligomycin sensitivity-conferring protein) vermittelt, das den F1-Kopf mit dem peripheren Schaft verbindet. Angesichts des hohen Grades an Sequenz- und Strukturkonservierung zwischen OSCP und der äquivalenten δ-Untereinheit von Chloroplasten und Bakterien über einen Zeitraum von ca. 1,5 Milliarden Jahren ist es wahrscheinlich, dass der gleiche Mechanismus für alle F-Typ-ATPasen gilt [2].
![Abb. 1: CryoEM-Struktur der Chloroplasten F1Fo ATP-Synthase. A, B: Zwei zueinander senkrechte Ansichten der 2,9 - 3,4 Å-Karte des monomeren Komplexes mit dem c-Ring (gelb) in der Membran (graues Band). Der F1-Kopf (grün) ist mit der Untereinheit a (hellblau) durch den flexiblen peripheren Schaft (violett) verbunden, der aus zwei langen, verschlungenen Helices besteht, die über die Untereinheit δ (orange) mit dem F1-Kopf verbunden sind. Der aus den Untereinheiten γ (blau) und ε (magenta) bestehende zentrale Stiel überträgt das Drehmoment vom c-Ring-Rotor auf F1. C, D: Gebundenes Adenosindiphosphat (ADP) in den katalytischen Bindungstaschen der Untereinheiten β mit gelösten Mg-Ionen und Wassermolekülen. ATP wurde während der Isolierung zu ADP hydrolysiert [3]. Abb. 1: CryoEM-Struktur der Chloroplasten F1Fo ATP-Synthase. A, B: Zwei zueinander senkrechte Ansichten der 2,9 - 3,4 Å-Karte des monomeren Komplexes](https://www.mpg.de/14493590/original-1591539546.jpg?t=eyJ3aWR0aCI6ODQ4LCJmaWxlX2V4dGVuc2lvbiI6ImpwZyIsIm9ial9pZCI6MTQ0OTM1OTB9--cd36f466adf32e949c6a968c5fc724f0c746a51e)
![Abb. 2: CryoEM-Struktur des Dimers der mitochondrialen ATP-Synthase aus Polytomella. A: Seitenansicht der 2,7 Å-Elektronendichte-Karte mit farbig dargestellten Untereinheiten auf der rechten Seite. Das Dimer biegt die Lipid-Doppelschicht (graues Band). Der massive Stator besteht aus zehn einzigartigen ASA-Untereinheiten (ATP synthase-associated proteins), darunter die von uns neu entdeckte Komponente ASA10 (rot), die das Dimer in der Membran auf der luminalen Seite zusammenhält. B: Die dreizehn Unterzustände der primären Rotationszustände 1, 2 und 3, die durch Bildverarbeitung und 3D-Klassifikation aufgelöst werden konnten. Untereinheit β, hellgrün; α, dunkelgrün; γ, blau; Stator grau [5]. Abb. 2: CryoEM-Struktur des Dimers der mitochondrialen ATP-Synthase aus Polytomella. A: Seitenansicht der 2,7 Å-Elektronendichte-Karte mit farbig darg](https://www.mpg.de/14493644/original-1591539546.jpg?t=eyJ3aWR0aCI6ODQ4LCJmaWxlX2V4dGVuc2lvbiI6ImpwZyIsIm9ial9pZCI6MTQ0OTM2NDR9--ca3d12140888f48de08baaab0930088fe4ead164)