Molekulare Mechanismen für die Formung und Qualitätskontrolle von Lipidmembranen
Forschungsbericht (importiert) 2017 - Max-Planck-Institut für Biophysik
Membranen als Zentren zellulärer Aktivität
Lebende Zellen sind von Membranen umhüllt, um sie vor unkontrollierten Einflüssen der Umgebung zu schützen. Auch das Innere von Zellen höherer Organismen wird durch Membranen unterteilt, zum Beispiel in das endoplasmatische Retikulum (ER), dessen Membran auch den Zellkern umhüllt und in ein feines Netzwerk von engen Membranröhren sowie flachen Membranbereichen strukturiert ist. Wie erhalten Membranen ihre oft ungewöhnliche Architektur und wie führen Zellen die Qualitätskontrolle von Membranen durch?
Qualitätskontrolle des endoplasmatischen Retikulums
Zelluläre Membranen bestehen aus einer Vielzahl unterschiedlicher Lipide sowie Cholesterin und Proteinen. Veränderungen in der Zusammensetzung können die Membran verdicken und deren Viskosität erhöhen. Um dem zu begegnen, kontrollieren Hefezellen den Sättigungsgrad des ER mithilfe des Proteins Mga2 [1]. Mga2 besitzt eine einzelne Transmembranhelix, die den Kern der sensorischen Aktivität bildet. Molekulardynamik-Simulationen dieser Helices in Membranen unterschiedlicher Sättigung zeigten, dass je zwei Helices mit unterschiedlichen, vom Sättigungsgrad der Lipide abhängigen Bindungsflächen dimerisieren. Ein Tryptophan-Rest, im Zentrum der Membran lokalisiert, wird in dichtgepackten Membranen hoher Sättigung ins Innere des Dimers gedrängt, in locker gepackten Membranen zeigt der Tryptophan-Rest nach außen.
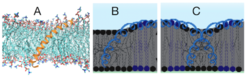
Ein weiterer wichtiger Sensor des ER ist Ire1 [2]. Dieses Protein wurde speziell zu seiner Funktion in der unfolded protein response untersucht, die der Bildung von gefährlichen Proteinaggregaten entgegenwirkt. Interessanterweise reagiert Ire1 aber auch auf sogenannten bilayer stress, etwa in der Form geänderter Lipidzusammensetzungen. Wiederum trugen Molekulardynamik-Simulationen wesentlich zum Verständnis des Sensors bei. Sie zeigten, dass der amphipathische Teil der Transmembranhelix von Ire1 nahezu flach in der Membran mit seiner hydrophoben Seite nach innen und der polaren Seite nach außen liegt (Abb. 1A). Der Grad lokaler Störungen in der Membran hängt von der Sättigung der Lipide ab und kann durch Cluster-Bildung insgesamt verringert werden (Abb. 1B, C).
Gestaltung der Membranarchitektur – Fusion
Auch die dreidimensionale Struktur der Membranen muss von Zellen gestaltet werden. Ein wesentlicher Prozess ist dabei die Fusion von Membranen, etwa beim Freisetzen von Neurotransmittern in Synapsen. Um die hohen energetischen Barrieren kontrolliert zu überwinden, finden komplexe Prozesse statt. Die Fusion von Membranen ist auch biotechnologisch von großem Interesse, etwa um Medikamente gezielt in Zellen zu transportieren. Noch ist wenig darüber bekannt, wie die Fusion von Membranen im Detail vonstattengeht und wie sie vielleicht beschleunigt werden kann. Molekulardynamik-Simulationen von Membranvesikeln, die mit Karbonnanoröhrchen miteinander verbunden sind, lieferten inzwischen neue Einsichten [3]. Die Simulationen zeigten, dass die Röhrchen die hohe kinetische Barriere nahezu vollständig beseitigen und zumindest kleine Vesikel zur spontanen Fusion anregen. Dabei gleiten die Lipide entlang der hydrophoben Röhrchen, was zu einer lokalen Störung der Membran führt, die wiederum die Durchmischung der Lipidschichten ermöglicht.
Gestaltung der Membranarchitektur – Tubuli im ER
Auch zur Bildung der außergewöhnlichen Struktur des Röhrchen-Netzwerks des ER lieferten rechnerische Untersuchungen neue Einsichten [4]. Gemeinhin wird angenommen, dass die Tubuli durch Proteine, insbesondere Retikulone, stabilisiert werden. Dennoch gibt der hohe energetische Aufwand für die Bildung der hochgekrümmten Membranen des ER immer noch Rätsel auf - man würde nämlich erwarten, dass sich die ER-Tubuli unter energetischen Gesichtspunkten spontan in flächige Strukturen umordnen müssten. Mithilfe von Simulationen konnten wir jedoch zeigen, dass die Tubuli im hohen Grade metastabil sind. Dies ermöglicht es, Röhrenstrukturen durch Einfügen von Lipiden in Membranvesikel zu induzieren, sofern deren Volumen reguliert wird. Zellen machen sich diese vorteilhafte Energetik zunutze, um so Röhrennetzwerke des ER zu erzeugen und zu erhalten.
Gestaltung der Membranarchitektur – Autophagie
Die metastabilen Tubuli sind hingegen ein Problem in der Bildung von Phagophoren. Das Aushungern induziert in Zellen höherer Organismen die Autophagie, bei der ganze Organellen als Nahrungsersatz abgebaut werden. Diese werden dabei von einer Doppelmembran umhüllt, die dann mit dem Lysosom fusioniert, wo der Inhalt des Phagophors abgebaut wird. In Hefezellen bildet sich das Phagophor durch die Fusion von Vesikeln. Ab drei fusionierten Vesikeln sollte die Schalenstruktur des Phagophors stabil sein. Bedingt durch die hohe Metastabilität der Tubuli verhindert jedoch eine hohe Barriere die spontane Bildung von Phagophoren [4, 5].
An der Bildung von Phagophoren ist auch ein sehr ungewöhnliches Protein beteiligt, der fast 40-nm lange, S-förmige Atg1-Dimerkomplex. Simulationen eines Modells dieser Proteine an der Membranoberfläche zeigten, dass die S-Form ideal dazu geeignet ist, die Barriere auf dem Weg zum Phagophor zu beseitigen [5]. Dies wurde durch Messungen in der Gruppe von James Hurley in lebenden Zellen bestätigt, bei denen geschwächte Membranwechselwirkungen die Bildung von Phagophoren verhinderten [5].
Insgesamt ermöglichten es molekulare und coarse-grained Simulationen, ein detailliertes Bild wichtiger Prozesse bei der Qualitätskontrolle von Membranen [1, 2] und deren räumlicher Gestaltung zu gewinnen [3-5].
Literaturhinweise
Molecular Cell 67, 63-684 (2017)
