Aufklärung molekularer Mechanismen des Membrantransports mithilfe von Elektronenmikroskopie und Röntgenkristallographie
Forschungsbericht (importiert) 2006 - Max-Planck-Institut für Biophysik
Die Abteilung Strukturbiologie am Max-Planck-Institut für Biophysik (Direktor: Werner Kühlbrandt) arbeitet an der Aufklärung der Struktur von Membranproteinen und Membranproteinkomplexen, mit dem Ziel, ihre molekularen Mechanismen zu verstehen. Für die Herstellung der Proteine werden molekularbiologische und biochemische Methoden (Klonierung, Expression in geeigneten heterologen Systemen, Reinigung) eingesetzt. Die gereinigten Proteine werden für Kristallisationsexperimente verwendet. Bei Membranproteinen, die nur im Maßstab von wenigen Milligramm isoliert werden können, steht die zweidimensionale (2D) Kristallisation im Vordergrund. Mit Membranproteinen, die in ausreichender Menge (>10 mg) anfallen, werden 3D-Kristallisationsexperimente durchgeführt.
3D-Kristalle werden für die Datensammlung mit Synchrotronstrahlen, vor allem an der Max Planck Beamline in Villingen (Schweiz), für die röntgenkristallographische Strukturbestimmung mit atomarer Auflösung eingesetzt. 2D-Kristalle werden zur elektronenmikroskopischen Strukturbestimmung im Auflösungsbereich von 3.5 bis 8 Ångström (Å) herangezogen. Die Struktur von Proteinkomplexen, die ausreichend groß sind (>300 kDa), aber nur in sehr geringen Mengen anfallen (<1 mg) oder sich nicht kristallisieren lassen, wird mithilfe der Einzelpartikelanalyse im Elektronenmikroskop bei 10-30 Å Auflösung untersucht. Darüber hinaus machen die Wissenschaftler die Organisation großer Membrankomplexe in nativen Membranen mithilfe der Elektronen-Cryotomographie sichtbar und arbeiten an der Entwicklung eines neuartigen, korrigierten Elektronenmikroskops für die Biologie.
Regulation der Stickstoff-Aufnahme in Bakterien und Archaeen
(Özkan Yildiz, Christoph Kalthoff, Stefan Raunser, Werner Kühlbrandt)
Während tierische Organismen Stickstoff in Form von Eiweiß oder Aminosäuren aufnehmen, erfolgt die Stickstoffaufnahme bei Prokaryonten und Pflanzen vorwiegend in Form von Ammoniak (NH3) oder Ammonium-Ionen (NH4+). Bis vor kurzem ist man davon ausgegangen, dass die Zellmembran dieser Organismen spezielle Ammonium-Transporter enthält, welche die geladenen Ammonium-Ionen durch die dafür undurchlässige Membran schleusen. Erst die Röntgenstruktur eines vermuteten Ammonium-Transporters einer kalifornischen Arbeitsgruppe im Jahr 2004 hat gezeigt, dass sehr wahrscheinlich nicht das Ammonium-Ion, sondern ungeladenes Ammoniak aufgenommen wird. Entsprechend handelt es sich bei dem Aufnahme-Protein nicht um einen aktiven Transporter, sondern um einen passiven Kanal.
Die Ammoniak-Aufnahme durch diesen Kanal muss streng reguliert werden, damit der intrazelluläre pH-Wert nicht zusammenbricht. Schon seit längerem ist bekannt, dass an der Regulation der Stickstoffaufnahme Signalproteine, die so genannten PII-Proteine, beteiligt sind, die auch an zahlreichen anderen Prozessen des Stickstoff-Stoffwechsels mitwirken und zu den einfachsten, ältesten und am höchsten konservierten Signalproteinen überhaupt gehören. Die Signalproteine erkennen anhand der Konzentration kleiner Stoffwechselprodukte wie ATP und 2-Ketoglutarat, ob die Zelle Stickstoff benötigt. Der zugrunde liegende Regulationsmechanismus war jedoch weitgehend unbekannt.
Um diesen Mechanismus besser zu verstehen, haben die Wissenschaftler um Werner Kühlbrandt den Ammoniak-Kanal Amt1 ebenso wie das dazugehörige PII-Signalprotein GlnK1 aus dem hyperthermophilen Archaeon Methanococcus jannaschii in E. coli exprimiert und gereinigt. Anschließend wurden die beiden Proteine zusammengegeben, wobei sie einen Komplex bildeten. Die Forscher konnten zeigen, dass die Komplexbildung nur in Abwesenheit der Effektor-Moleküle ATP und Ketoglutarat vollständig verläuft. Der stabile Komplex wurde dann im Elektronenmikroskop abgebildet. Es zeigte sich, dass ein Trimer des Ammoniak-Kanals Amt1 auf der cytoplasmatischen Seite ein Trimer des Signalproteins bindet (Abb. 1). Inzwischen wurde die Röntgenstruktur homologer Komplexe von zwei konkurrierenden Gruppen bestimmt.
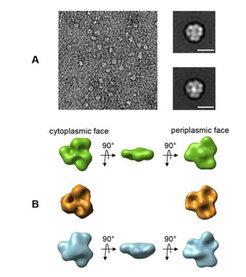
Die Komplexstruktur selbst sagt jedoch nichts über den Regulationsmechanismus aus, sondern zeigt lediglich, dass drei Peptidschleifen des Signalprotein-Trimers in die drei Poren des Ammoniak-Kanals hineinragen und diese schließen, was die Forscher schon ihrer elektronenmikroskopisch bei niedriger Auflösung bestimmten Komplex-Struktur entnehmen konnten.

Zum Verständnis des Mechanismus war es notwendig, die Struktur des Signalproteins GlnK1 in drei verschiedenen Zuständen zu bestimmen, und zwar (1) ohne gebundene Effektoren, (2) mit gebundenem Mg-ATP sowie (3) mit gebundenem Mg-ATP und Ketoglutarat, was auch gelang. In Abwesenheit der Effektoren sind die Peptidschleifen des Signalproteins ausgeklappt und flexibel (Abb. 2A), sodass sie leicht in die Kanalporen eindringen und diese versiegeln. Dieser Vorgang wird offensichtlich durch Ladungskomplementarität zwischen den Peptidschleifen (positiv geladen) und den Ausgängen der Porenkanäle (negativ geladen) befördert (Abb. 3).

In Gegenwart von Mg-ATP nehmen die Peptidschleifen eine kompakte Konformation ein (Abb. 2B), so dass sie kaum mehr in die Kanalpore eindringen können. Dieser Befund deckt sich mit den biochemischen Experimenten, die zeigten, dass Mg-ATP die Komplexbildung weitgehend verhindert.
Vollständig unterbunden wird sie erst durch Zugabe von Ketoglutarat, und die dritte Struktur macht deutlich, warum das so ist: In der durch Mg-ATP fixierten Form entsteht eine Bindungsstelle für dieses bei neutralem pH doppelt negativ geladene Molekül, es bindet genau an der Spitze der eingeklappten Peptidschleife und fixiert die kompakte Form zusätzlich (Abb. 2C). Wegen Ladungsabstoßung kann sich der Komplex mit dem Kanalprotein nicht bilden. Interessanterweise fanden die Wissenschaftler am Frankfurter MPI nur eine der drei Bindungsstellen im Trimer durch Ketoglutarat besetzt, in exakter Übereinstimmung mit älteren biochemischen Daten einer amerikanischen Gruppe, die zeigten, dass ein Molekül Ketoglutarat pro Trimer genügt, um die Stickstoffaufnahme zu gewährleisten. Niedrige Konzentrationen von Ketoglutarat und ATP signalisieren, dass die Zelle keinen Bedarf an Stickstoff hat. Die gebundenen Effektor-Moleküle fallen vom Signalprotein ab, die Peptidschleifen klappen aus und versiegeln die Ammoniak-Poren. Mit diesem einfachen molekularen Rückkopplungsmechanismus wird die Stickstoffzufuhr reguliert [1].
pH-induzierte Konformationsänderung von Natrium-Protonen-Austauschproteinen
(Kutti Ragunath Vinothkumar, Matthias Appel, Werner Kühlbrandt)
So gut wie alle Organismen verwenden Natrium-Protonen-Austauschproteine in der Zellmembran zur genauen Einstellung und Regulierung des intrazellulären pH-Werts, einem der zentralen Parameter für alle Lebensvorgänge. Sie wirken gleichzeitig als pH-Sensoren und als Antiporter, die die Na+-Ionen bzw. Protonen je nach Bedarf gegen einen Konzentrationsgradienten durch die Membran schleusen. Besonders gut untersucht ist das Natrium-Protonen-Austauschprotein NhaA aus E. coli, das bei niedrigem intrazellulärem pH-inaktiv ist und seine maximale Aktivität bei pH 8 erreicht. Seine Röntgenstruktur im inaktiven Zustand wurde vor kurzem in der Nachbarabteilung von Hartmut Michel bestimmt. Die Plasmamembran von Säugerzellen enthält eine Reihe verschiedener, vermutlich ähnlich gebauter Austauschproteine, z.B. NHE1, deren heterologe Expression jedoch problematisch ist, sodass diese für Strukturuntersuchungen bisher nicht in Frage kommen. Eine Stellung zwischen Bakterien und höheren Organismen nehmen die Austauschproteine von Archaeen ein, die sich leichter herstellen und kristallisieren lassen als ihre eukaryontischen Verwandten, zu denen sie aber funktionell ähnlicher zu sein scheinen als zu ihren bakteriellen Homologen.
Das Team um Werner Kühlbrandt hat das Natrium-Protonen-Austauschprotein NhaP1 aus dem thermophilen Archaeon Methanococcus jannaschii in E. coli exprimiert und kristallisiert. Die Elektronenkristallographie von 2D-Kristallen ergab, dass MjNhaP1 wie NhaA in der Membran Dimere bildet. Auch die Form der beiden Austauschproteine ist ähnlich, ein genauer Vergleich der Projektionskarten zeigt jedoch signifikante Unterschiede (Abb. 4). Interessanterweise verhält sich dieses Protein genau umgekehrt wie NhaA, das heißt es ist bei neutralem pH inaktiv und erreicht seine volle Aktivität erst bei pH 6 oder darunter.

Die pH-Aktivierung des Proteins ebenso wie der Transportvorgang setzen eine Konformationsänderung voraus. Um den Aktivierungs- bzw. Transportvorgang zu verstehen, muss die Struktur der verschiedenen Konformationen bekannt sein. Bei dreidimensionalen Kristallen ist es schwierig, Konformationsänderungen auszulösen, ohne die Kristalle zu zerstören. Durch Inkubation von 2D-Kristallen von NhaA und NhaP1 bei verschiedenen pH-Werten konnten die Strukturbiologen jedoch die Konformationsänderungen mithilfe der Elektronenkristallographie unmittelbar sichtbar machen. Bei NhaP1 ändert eine Transmembranhelix ihre Lage (Abb. 4B), was vermutlich mit der Aktivierung des Austauscher-Proteins zusammenhängt [2]. Bei NhaA konnten zwei verschiedene Konformationsänderungen beobachtet werden, von denen eine pH-induziert war und das Protein aktivieren dürfte, während die andere durch Na-Ionen induziert wurde und sehr wahrscheinlich den Ionentransport durch die Membran bewirkt.
Struktur und Schaltfunktion eines monomeren Porins
(Özkan Yildiz, Kutti Ragunath Vinothkumar, Werner Kühlbrandt)
Kleine Moleküle (bis einige 100 Da) diffundieren nahezu ungehindert durch porenbildende Proteine in der Außenmembran von Bakterien, Mitochondrien und Chloroplasten. Diese so genannten Porine haben die charakteristische Struktur eines zylindrischen β-Faltblatts, das eine wässrige Pore umschließt. Alle porenbildenden Außenmembranproteine mit bisher bekannter Röntgenstruktur sind Trimere. Eine Ausnahme schien das Porin OmpG aus E. coli zu sein, von dem die Wissenschaftler auf Grund früherer Untersuchungen zweidimensionaler Kristalle annahmen, dass es monomere Poren bildet. Einzelkanalmessungen einer anderen Gruppe in Oxford zeigten eine weitere ungewöhnliche Eigenschaft: Anders als die bekannten trimeren Porine ist die Pore von OmpG unter physiologischen Bedingungen pH-reguliert, d.h. offen bei pH über 6 und darunter geschlossen.
Es gelang, OmpG, das normalerweise in E. coli nicht exprimiert wird, in großen Mengen als Einschlusskörper herzustellen, in vitro zu falten und das renaturierte Protein sowohl bei saurem als auch neutralem pH-Wert zu kristallisieren. Die Struktur (Abb. 5) zeigt, dass OmpG in der Tat ein monomeres, zylindrisches Porenprotein ist.

Im Gegensatz zu den klassischen trimeren Außenmembranporinen wird der Zylinder von 14 statt 16 oder 18 β-Strängen gebildet. Seine Architektur ist damit besonders einfach, ökonomisch und robust. Auf der nach außen gewandten Seite der Membran zeigt OmpG einige längere Peptidschleifen. Bei neutralem pH ist die Pore offen (Abb. 5A und B), bei niedrigem pH lagert sich die längste Peptidschleife um und schließt die Pore (Abb. 5C). Der Schalter für die Umlagerung ist offensichtlich ein Histidin-Paar, das bei neutralem pH-Wert Wasserstoffbrücken untereinander und mit benachbarten Seitenketten ausbildet und zwei nebeneinander liegende β-Stränge stabilisiert. Bei sinkendem pH-Wert werden die Histidine positiv geladen und stoßen sich ab, wodurch die Schleife destabilisiert wird und sich umlagern kann [3].
Ein monomeres, stabiles Porin, das leicht in großen Mengen hergestellt werden kann und dessen Kanaleigenschaften auf einfache Weise schaltbar sind, ist für die Biosensor-Entwicklung attraktiv. Auf der Grundlage der in Frankfurt bestimmten Struktur versuchen die Forscher, die Schalteigenschaften zu modifizieren, sodass beispielsweise Schwermetallionen erkannt werden. Diese Entwicklungen wurden zum Patent angemeldet.
Eine Phasenplatte für die Elektronenmikroskopie
(Bastian Barton, Rasmus Schröder, Werner Kühlbrandt)
In der Lichtmikroskopie ist der Einsatz einer Phasenplatte zur Verbesserung des Kontrastes bei der Abbildung ungefärbter biologischer Objekte seit Jahrzehnten selbstverständlich. In der Elektronenmikroskopie, die mit noch geringerem Eigenkontrast biologischer Strukturen zu kämpfen hat, konnten Phasenplatten bisher nicht angewendet werden, obwohl die Theorie für das Elektronenmikroskop bereits 1947 durch den deutschen Elektronenoptiker Hans Boersch entwickelt wurde. Das liegt daran, dass Phasenplatten vom Boersch-Typ eine elektrostatische Einzellinse mit Innendurchmesser von circa 1 µm erfordern. Solche miniaturisierten elektronenoptischen Elemente können erst jetzt mithilfe von Fertigungstechniken der Nanotechnologie erzeugt werden, insbesondere die Mikrostrukturierung sowie Ionenstrahl-Bearbeitungsverfahren zum präzisen Bohren kleinster Öffnungen in Metallfolien. Mit diesen Verfahren wurde in Zusammenarbeit mit der Arbeitsgruppe Gerthsen an der Technischen Universität Karlsruhe eine entsprechende fünfschichtige Einzellinse entwickelt und hergestellt (Abb. 6) und in ein Elektronenmikroskop eingebaut. Durch Anlegen einer variablen Spannung an die elektrostatische Einzellinse konnte die Phase des Primärstrahls in einem Bereich von 00 bis etwa 2000 verschoben werden. Für maximalen Phasenkontrast ist eine Phasenschiebung von 900 erforderlich. Somit konnten die Wissenschaftler erstmals die Funktion der Boersch-Phasenplatte im Elektronenmikroskop nachweisen [4].

Für die praktische Anwendung sind weitere Entwicklungen erforderlich. So ist es notwendig, das erste Beugungsbild in der Brennebene der Objektivlinse um einen Faktor 5-10 zu vergrößern, um die Abschattung durch die Linse und die Trägerstege zu minimieren. Darüber hinaus lässt die Kombination der Boersch-Phasenplatte mit einem Korrektor der chromatischen Aberration der Objektivlinse (elektronenoptischer Achromat) für alle Bereiche der Elektronen-Cryomikroskopie biologischer Objekte, einschließlich der Einzelpartikel-Abbildung und der Elektronentomographie optimale, bisher unerreichbare Abbildungsbedingungen erwarten. Die Entwicklung eines korrigierten Elektronenmikroskops mit Phasenplatte durch das MPI für Biophysik gemeinsam mit den Firmen Zeiss NTS (Oberkochen) und CEOS (Heidelberg) war wesentlicher Bestandteil des Antrages auf einen Exzellenzcluster zum Thema „Macromolecular Complexes“ der Johann Wolfgang Goethe-Universität in Frankfurt am Main, der von der DFG seit dem 1.11.2006 gefördert wird. Die Entwicklungsarbeiten haben bereits begonnen und die Beteiligten gehen davon aus, dass das korrigierte Elektronenmikroskop in drei bis vier Jahren einsatzbereit sein wird.





