Der Ein-Kohlenstoff-Überträger Tetrahydromethanopterin in Enzymen
Forschungsbericht (importiert) 2004 - Max-Planck-Institut für Biophysik
Tetrahydromethanopterin (H4MPT)
Bei einer Reihe von anabolen und katabolen Stoffwechselprozessen ist die Übertragung von C1-Einheiten von essentieller Bedeutung. Diese C1-Einheiten werden meist über Stickstoff- oder Schwefelatome kovalent an niedermolekulare organische Verbindungen in einer aktivierten Form und in den Oxidationsstufen von Kohlendioxid, Formiat, Formaldehyd und Methanol gebunden. Der vielseitigste C1-Überträger ist das Tetrahydrofolat (H4F) (Abb. 1), das C1-Einheiten in Form von Formyl-, Methenyl-, Formimino-, Methylen- und Methyl-Grupppen binden, aktivieren und transferieren kann. Für biosynthetische Methylierungen, die ein hohes Transferpotenzial verlangen, verwendet die Natur das reaktivere S-Adenosylmethionin, für die Übertragung von CO2 Biotin. Manche Mikroorganismen verwenden für speziellere Stoffwechselwege weitere C1-Überträger. Von besonderem Interesse ist dabei das mit H4F strukturell nahe verwandte H4MPT, das C1-Einheiten in denselben Oxidationsstufen binden kann (Abb. 1). Nach seiner Entdeckung vor fast 25 Jahren [1, 2] glaubte man lange Zeit, H4MPT käme nur in methanogenen Archaebakterien, kurz Archaeen vor, wo es einen essentiellen Cofaktor für eine Reihe von Enzymen darstellt, die bei der Methanbildung aus H2 und CO2 beteiligt sind [3]. Die größere Verbreitung von H4MPT wurde erst im letzten Jahrzehnt evident. Zunächst wurde H4MPT in Sulfat reduzierenden Archaeen entdeckt, die phylogenetisch verwandt zu methanogenen Archaeen sind [4].
Überraschender war dann, dass H4MPT sogar außerhalb von Archaeen als lebenswichtiger Cofaktor im zentralen Metabolismus von vielen methylotrophen α-, β- und χ-Proteobakterien vorkommt [5]. Erst vor kurzem wurde nachgewiesen, dass nicht nur die aerobe Methanoxidation, sondern auch die anaerobe Methanoxidation H4MPT-abhängig ist [6]. Diese wird von Archaeen durchgeführt, die verwandt sind zu den Methanosarcinalen (Untergruppe der methanogenen Archaeen). Alle diese Organismen sind auf den C1-Stoffwechsel spezialisiert und von großer ökologischer Bedeutung im globalen Kohlenstoffmetabolismus. Die kürzliche Entdeckung von H4MPT-abhängigen Enzymen in der rätselhaften Bakteriengruppe der Planctomyceten eröffnete die Debatte über die Evolution der H4MPT und H4MPT-abhängigen Enzyme von Neuem, weil phylogenetische Untersuchungen die Planctomyceten gleich weit von den Archaeen wie von den Proteobakterien platzieren [7].
Der größte funktionell relevante Unterschied zwischen den Strukturen von H4MPT and H4F liegt in der Elektronen schiebenden Methylengruppe des H4MPT in Position 1c (Abb. 1), die mit dem N10-Atom durch den aromatischen Ring verbunden ist, wohingegen H4F eine Elektronen ziehende Carbonylgruppe in dieser Position hat. Aus diesem Grund ist das Redoxpotential des Methenyl-H4MPT+/Methylen-H4MPT-Paares (-390 mV) und des Methylen-H4MPT+/Methyl-H4MPT-Paares (-310 mV) fast 100 mV negativer als die der entsprechenden H4F-Paare.
H4F und H4MPT sind höchstwahrscheinlich Produkte einer konvergenten Evolution. So unterscheiden sich die Biosynthesewege von H4F und H4MPT und die daran beteiligten Enzyme signifikant. Ferner deuten Sequenz- und Strukturvergleiche auf eine separate Entwicklung von H4MPT- und H4F-abhängigen Enzymen hin, die ähnliche Reaktionen katalysieren. Interessanterweise sind die meisten Enzyme, die die Umwandlung von C1-Derivaten katalysieren, hochspezifisch für entweder H4MPT oder H4F.
Bisher wurden sieben H4MPT-abhängige Enzyme strukturell aufgeklärt. Dabei gestaltete sich die Bindung von H4MPT oder eines seiner Derivate an die Enzyme im kristallinen Zustand als sehr schwierig (übrigens wie die Bindung von H4F an H4F-abhängige Enzyme), sodass eine strukturelle Charakterisierung von H4MPT in einem Enzym erst kürzlich mit dem Formaldehyd- aktivierenden Enzym (Fae)-Methylen-H4MPT-Komplex und dem Formylmethanofuran: H4MPT formyltransferase (Ftr)-H4MPT-Komplex gelang. Bei diesen Arbeiten kooperierten die Wissenschaftler am MPI für Biophysik eng mit den Arbeitsgruppen von Rudolf K. Thauer, Seigo Shima und Julia A. Vorholt (Max-Planck-Institut für terrestrische Mikrobiologie in Marburg).
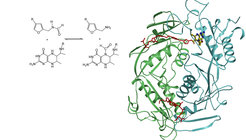
Der Fae-Methylen-H4MPT-Komplex
Fae ist ein Enzym des Energiemetabolismus und der Formaldehyd-Detoxifikation des methylotrophen Proteobakteriums Methylobacterium extorquens AM1 und katalysiert die Bildung von Methylen-H4MPT aus H4MPT und Formaldehyd (Abb. 2). Fae stellt einen homopentameren Proteinkomplex (Mr 18 kDa per Monomer) dar, der in drei Schichten unterteilbar ist [8]. Der innere α-helikale Ring besteht aus fünf miteinander verknüpften α-Helices, der äußere Ring aus drei α-Helices. Der zentrale Ring besteht aus fünf β-Faltblättern mit jeweils fünf Strängen. Die Bindungsstelle für Methylen-H4MPT liegt in einer Spalte zwischen den Untereinheiten, in die der Cofaktor in einer „S“- Form eingebettet ist (Abb. 1b). Das Protein hält Methylen-H4MPT im Wesentlichen nur an seinem Pterin- und Benzylteil durch Wasserstoffbrückenbindungen beziehungsweise hydrophobe Wechselwirkungen fest. Die Bindung von Methylen-H4MPT induziert Seitenkettenverschiebungen von bis zu 5 Å, eine starre Konformation in dem zuvor flexiblen C-terminalen Arm, die Ausbildung eines neues hydrophoben Bereichs und die Inversion der Amidgruppe einer Glutamin-Seitenkette. Nur die neue Konformation der Seitenkette erlaubt die Knüpfung von zwei Wasserstoffbrücken zum Pyrimidinring des Methylen-H4MPT. Die Formaldehydkondensation an H4MPT erfolgt durch einen nukleophilen Angriff des N5-Atoms von H4MPT auf den Kohlenstoff des Formaldehyds. Diese Reaktion wird optimal durch ein Histidin unterstützt, welches ein Proton auf das entstehende Carbonylanion übertragen kann.

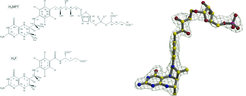
Der Ftr-Formylmethanofuran-H4MPT-Komplex
Ftr ist ein Enzym im Energiemetabolismus und bei der autotrophen CO2-Fixierung von methanogenen und Sulfat-reduzierenden Archaeen. Bei methylotrophen Bakterien ist es an der Methanoloxidation zu CO2 beteiligt. Ftr katalysiert reversibel die Übertragung einer Formylgruppe von Formylmethanofuran auf H4MPT (Abb. 3). Das Enzym liegt als Homotetramer (Mr 35 kDa per Monomer) vor, das sich treffender als Dimer aus zwei Dimeren beschreiben lässt. Die Substratbindungsstellen für Methanofuran und H4MPT liegen zwischen zwei eng verzahnten Monomeren der einzelnen Dimeren [9]. Während Methanofuran in einer tiefen, an die Struktur des Cofaktors angepassten Spalte zwischen den Monomeren bindet, ist die H4MPT-Bindungsspalte relativ weit und flach. Der Besetzungsgrad von H4MPT ist gering, wobei der Pterinteil, jedoch nicht der Rest des Moleküls, klar sichtbar ist. Der Pterinring ist vollständig vom Lösungsmittel abgeschirmt und seine polaren Atome sind mit konservierten Resten der Polypeptidkette verknüpft. Die Phenylgruppe ist dagegen von hydrophoben und aromatischen Resten umgeben. Trotz der beträchtlichen Größe der Substrate induziert ihre Bindung keine markanten Konformationsänderungen am Protein. Beide Substratbindungsspalten sind an ihrem Ende im Inneren des Proteins verbunden und genau dort befindet sich das aktive Zentrum. Eine besondere Funktion kommt einem streng konservierten Glutamat zu, das aufgrund seiner hydrophoben Umgebung und seiner Solvensabgeschirmtheit vermutlich protoniert ist. Es ist sowohl mit dem Pterinring als auch mit der Formylgruppe des Formylmethanofurans durch eine Wasserstoffbrückenbindung verknüpft und könnte das erwähnte Proton im Übergangszustand auf die Formylgruppe übertragen, nachdem der Formylkohlenstoff vom N5-Atom des H4MPT nukleophil angegriffen wurde.