Struktur und molekulare Mechanismen von Membran-Transport-Proteinen
Forschungsbericht (importiert) 2003 - Max-Planck-Institut für Biophysik
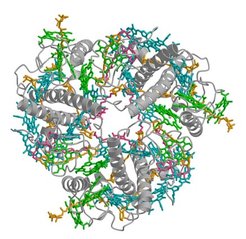
Nach mehr als 20-jähriger Arbeit ist es der Arbeitsgruppe um Werner Kühlbrandt gelungen, die Struktur des Lichtsammlerkomplexes (LHC-II) aus Erbsen-Chloroplasten zu bestimmen. LHC-II ist das hauptsächliche Antennenprotein in grünen Pflanzen und damit das häufigste Membranprotein auf der Erde. Seine Aufgabe ist es, die Sonnenenergie für die Photosynthese einzufangen und an das Reaktionszentrum des Photosystems II weiterzuleiten. LHC-II bildet kleine, sehr dünne 3-D-Kristalle, die schwer einzufrieren waren. Nur zwei von mehreren hundert untersuchten Kristallen waren in allen Raumrichtungen gut geordnet. Die Struktur wurde durch molekularen Ersatz mit der 1994 von der Arbeitsgruppe veröffentlichten, elektronenmikroskopisch bestimmten 3.4 Å-Dichtekarte gelöst und bis auf 2.5 Å verfeinert.
LHC-II ist ein Trimer, das aus drei identischen oder nahezu identischen Untereinheiten besteht (siehe Abb. 1). Jede Untereinheit enthält 14 Chlorophylle (8 Chlorophyll a, 6 Chlorophyll b), 4 Carotinoide (2 Luteine, ein Neoxanthin, ein Violaxanthin) und zwei Lipide. Die Chlorophylle sind so angeordnet, dass die Anregungsenergie verlustfrei von einem zum nächsten übergeht. Die Anordnung der Carotinoide im Komplex zeigt, dass sie sowohl eine Antennen- als auch eine Schutzfunktion haben. Die konjugierten π-Systeme der beiden Lutein-Moleküle im Zentrum des Monomers liegen exakt parallel zu denen der benachbarten Chlorophylle und sind so ausgerichtet, dass sie die gefährlichen Chlorophyll-Triplett-Zustände unschädlich machen.
Das Violaxanthin-Molekül ist an einem weiteren wichtigen Schutzmechanismus beteiligt. Es ist im Komplex vergleichsweise schwach gebunden und leicht austauschbar. Wenn der pH-Wert auf der lumenalen Seite der Thylakoidmembran bei hohem Lichteinfall und entsprechend hoher photosynthetischer Aktivität auf ungefähr 5 sinkt, wird ein Enzym angeschaltet, das die Epoxidgruppen an beiden Enden dieses Carotinoids entfernt. Das dabei entstehende Carotinoid Zeaxanthin bindet stärker an die gleiche Bindungsstelle und verdrängt noch vorhandenes Violaxanthin. Das π-System von Zeaxanthin ist um zwei konjugierte Doppelbindungen länger als das von Violaxanthin und liegt energetisch niedriger. Deswegen wird die von den Chlorophyllen eingefangene Energie in diesem Fall statt auf benachbarte Komplexe auf das Zeaxanthin übertragen und geht in Form von Wärme verloren. So wird verhindert, dass zu große Sonneneinstrahlung irreversible photochemische Schäden an den Antennen- und Reaktionszentren-Komplexen der Photosynthesemembran verursacht.
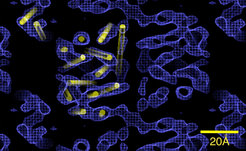
Kaliumkanäle spielen bei der Reizleitung und Signalverarbeitung in den Neuronen des Gehirns eine zentrale Rolle. Zwar kennt man inzwischen die Struktur von einfach gebauten bakteriellen und archaealen Ionenkanälen. Über die genaue Struktur und die Wirkungsmechanismen der weitaus wichtigeren neuronalen Kalium-Kanäle weiß man jedoch noch wenig. Ein Problem stellt bereits die große Vielfalt der Kanalproteine dar. Es gibt hunderte verschiedene, biochemisch sehr ähnliche Typen, die kaum zu trennen und daher für Strukturuntersuchungen unzugänglich sind. Zur Lösung dieses Problems ist es notwendig, einen einzelnen reinen Typ zu exprimieren, was bei eukaryontischen Membranproteinen schwierig ist. Mitarbeitern der Abteilung ist es gelungen, durch Ko-Expression der α- und β-Untereinheit des KV1.2-Kaliumkanals aus Rattenhirn das funktionale Kanalprotein als α4β4-Komplex im Milligramm-Maßstab herzustellen und zu isolieren. Die Struktur des α4β4-Komplexes wurde zunächst durch Einzelpartikel-Analyse im Elektronenmikroskop bestimmt (siehe Abb. 2). 2-D-Kristallisationsexperimente waren erfolgreich und die elektronenkristallographische Strukturbestimmung dieses Kaliumkanals ist im Gang.

Zu den faszinierendsten Membranproteinen gehören die, die große Proteinmoleküle durch die Membran transportieren. In der Abteilung "Strukturbiologie" untersuchen die Wissenschaftler zwei verschiedene Protein-Translokasen: den bakteriellen Sec-Translokase-Komplex und die Translokasen der äußeren und inneren Mitochondrienmembran (TOM/TIM).
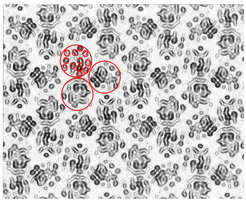
Der Sec-Komplex ist die universelle Proteinexport-Maschine in Bakterien. Er zeichnet sich dadurch aus, dass er Proteine transportiert, die für die Sekretion aus der Zelle bestimmt sind, aber auch Membranproteine in die innere Zellmembran einbaut. Durch Koexpression der drei Untereinheiten SecY, E und G konnten Mitarbeiter der Abteilung ausreichende Mengen des heterotrimeren SecYEG-Komplexes herstellen und mit diesem Material 2-D-Kristalle erhalten. Die 8 Å-Dichtekarte (siehe Abb.3) zeigt ein Dimer aus zwei Heterotrimeren. In jedem Heterotrimer lassen sich 15 membrandurchspannende Helices unterscheiden, von denen 10 zu SecY, 3 zu SecE und 2 zu SecG gehören. Die Zuordnung der Helices wurde möglich, als unter Mitwirkung von Mitarbeitern der Abteilung in den Arbeitsgruppen von Stephen C. Harrison und Tom A. Rapoport (Harvard Medical School) die Röntgenstruktur des homologen SecYEβ-Komplexes aus Methanococcus jannaschii gelöst wurde. Ein genauer Vergleich der Röntgenstruktur mit der 8 Å-Dichtekarte von SecYEG in der Membran zeigt interessante Unterschiede in der Lage und Orientierung der Helices, die Hinweise auf das erste Stadium des Transportmechanismus geben.
Die Protein-Translokase der äußeren Mitochondrienmembran (TOM-Komplex) besteht aus mindestens 6 verschiedenen Membranprotein-Untereinheiten. Die heterologe Überexpression des ganzen funktionsfähigen Komplexes ist bisher nicht möglich. Für biophysikalische und Strukturuntersuchungen bleibt daher nur die Isolation des TOM-Komplexes aus Mitochondrien. Da auf diese Weise nur vergleichsweise geringe Mengen hergestellt werden können, der Komplex aber circa 400 kDa (Kilo Dalton) groß ist, stellt die Einzelpartikel-Elektronenmikroskopie die beste Methode für eine erste Strukturbestimmung dar.
Die Komplexe werden in der Arbeitsgruppe Nikolaus Pfanner an der Universität Freiburg isoliert. Eine Dichtekarte des TOM-Komplexes (siehe Abb. 4a) zeigt zwei Translokationsporen. Ein TIM-Komplex der inneren mitochondrialen Membran (siehe Abb. 4b) hat ebenfalls zwei Poren, was auf einen räumlich koordinierten Transportmechanismus durch beide Komplexe hinweist.

Neben den genannten Transportsystemen befassen sich die Mitarbeiter der Abteilung "Strukturbiologie" mit der Struktur und Funktion von Sekundärtransportern, darunter dem osmoregulierten Betaintransporter Bet P, dem Natrium-Protonen-Austauscher NhaP und den Glutamat-Transportern Glt-P und GLT-I.
Diese Transporter wurden exprimiert. Von BetP (siehe Abb. 5) und NhaP wurden 2-D- beziehungsweise 3-D-Kristalle erhalten, mit denen die Wissenschaftler an der Strukturaufklärung arbeiten.
Literatur
[1] Parcej, D. N. and L. Eckhardt-Strelau: Structural characterization of neuronal voltage-sensitive K+ channels heterologously expressed in Pichia pastoris. Journal of Molecular Biology 333, 103-116 (2003).
[2] Breyton, C., W. Haase, T. A. Rapoport, W. Kühlbrandt and I. Collinson: Three-dimensional structure of the bacterial protein-translocation complex SecYEG. Nature 418, 662-665 (2002).
[3] Rehling, P., K. Model, K. Brandner, P. Kovermann, A. Sickmann, H. E. Meyer, W. Kühlbrandt, R. Wagner, K. N. Truscott and N. Pfanner: Protein insertion into the mitochondrial inner membrane by a twin-pore translocase. Science 299, 1747-1751 (2003).
[4] Model, K., T. Prinz, T. Ruiz, M. Radermacher, T. Krimmer, W. Kühlbrandt, N. Pfanner and C. Meisinger: Protein translocase of the outer mitochondrial membrane: role of import receptors in the structural organization of the TOM complex. Journal of Molecular Biology 316, 657-666 (2002).
[5] Ziegler, C., S. Morbach, D. Schiller, R. Krämer, C. Tziatzios, D. Schubert and W. Kühlbrandt: Projection structure and oligomeric state of the osmoregulated sodium/glycine betaine symporter BetP of corynebacterium glutamicum. Journal of Molecular Biology 337, 1137-47 (2004).




