Von der genetischen Information zur Behandlung von Krankheiten
Forschungsbericht (importiert) 2005 - Max-Planck-Institut für Biophysik
Genomische Sequenzierung
Die Basis aller molekularen Untersuchungen biologischer Prozesse im Menschen ist die Sequenz seines Genoms, die letztes Jahr in weitgehend endgültiger Form unter Beteiligung der Abteilung Analyse des Vertebratengenoms am Max-Planck-Institut für molekulare Genetik als einem von 22 Zentren fertig gestellt wurde [1]. Diese Sequenz hat unser Bild der biologischen Prozesse im Menschen dramatisch verändert. So stellte sich heraus, dass die gesamte Komplexität biologischer Prozesse im Menschen nur durch 20.000-25.000 Gene gesteuert wird, die für Eiweiße kodieren, sowie eine Anzahl von Genen, die direkt über ihre RNA wirken.
Mehr als 99 Prozent der euchromatischen (genreichen) Erbsubstanz des Menschen sind jetzt mit einer Genauigkeit von 99,999 Prozent entziffert. Im Gegensatz zur Rohfassung aus dem Jahr 2001 mit etwa 150.000 Lücken sind die 3 Milliarden Bausteine der humanen Sequenz nur noch an 341 Stellen unterbrochen. Diese Lücken liegen meist entweder in den für heutige Technologien unzugänglichen heterochromatischen (genarmen) Bereichen oder in Abschnitten, die im Genom mehrfach vorkommen. Damit haben sich die kontinuierlich zu lesenden Sequenzabschnitte von durchschnittlich 81.500 Basen auf 38,5 Millionen Basen, also um das 475fache, erhöht. Durch diese massive Verbesserung sind nicht nur weitaus genauere Studien über die Gene selbst möglich, sondern auch über die Bereiche, die regulatorische Funktion haben. Die Genauigkeit, mit der die Sequenz nun vorliegt, lässt aber auch erkennen, dass 58 Prozent aller Genvorhersagen von 2001 fehlerhaft waren [2, 3].
Die genetischen Ursachen vieler Erkrankungen konnten bereits identifiziert werden. Anhand der nun vorliegenden, nahezu vollständigen Sequenz können in Zukunft mit großer Sicherheit auch genetische Veränderungen, die komplexen Erkrankungen wie Krebs, Bluthochdruck, chronischen Entzündungen und Fettsucht zugrunde liegen, identifiziert werden. Auch wenn dieses Wissen zurzeit noch keine direkte Therapieanwendung für die Betroffenen bietet, so ist es doch die notwendige Voraussetzung für die Entwicklung zukünftiger gezielter Heilmöglichkeiten.
Die Abteilung Analyse des Vertebratengenoms war insbesondere an der Sequenzierung des Chromosoms 21 beteiligt, des zweiten menschlichen Chromosoms, dessen Sequenz mit extrem hoher Genauigkeit fertig gestellt werden konnte [4]. Die Evolution dieses Chromosoms sowie die molekulare Analyse der auf diesem Chromosom kodierten Gene stellt einen wichtigen Fokus der Arbeit der Abteilung dar.
Die Evolution von DNA-Sequenzen
Die Evolution der genomischen Sequenz gibt uns wichtige Anhaltspunkte über genetische Mechanismen, die für die Unterschiede zwischen den verschiedenen Spezies verantwortlich sind. Besonders interessant ist der Vergleich zwischen dem Genom des Menschen und seinem nächsten Verwandten, dem Schimpansen Pan troglodytes. Während sich die vergleichende Genomforschung zwischen relativ weit voneinander entfernten Organismen (evolutionäre Distanz zwischen Mensch und Maus: 60 Millionen Jahre) darauf konzentriert, Übereinstimmungen zwischen den Arten zu finden, führt der Vergleich mit dem Schimpansengenom (evolutionäre Distanz: etwa fünf bis sechs Millionen Jahre) zu greifbareren Unterschieden.
Ziel unserer Arbeit ist es, die molekulare Basis jener evolutionären Veränderungen zu entschlüsseln, die zu zwei Organismen mit deutlichen Unterschieden im Phänotyp und Verhalten geführt haben. Dadurch hoffen wir, nicht nur Informationen über die Funktion von Genen und regulatorischen Sequenzbereichen zu erhalten, sondern auch etwas über die biologischen Mechanismen zu lernen, die für Eigenschaften verantwortlich sind, welche den Menschen von anderen Lebewesen unterscheiden, wie z. B. Intelligenz und Sprache. Darüber hinaus wollen wir herausfinden, welche genetischen Unterschiede zur veränderten Empfänglichkeit für Krankheiten geführt haben.
Aus diesem Grund haben wir in einer asiatisch-deutschen Zusammenarbeit zusätzlich zu dem Chromosom 21 des Menschen auch das äquivalente Schimpansenchromosom 22 mit bisher unerreichter Genauigkeit sequenziert, analysiert und verglichen [5, 6]. Dabei stellten wir fest, dass die Zahl der geringfügigen Abweichungen - wenn also eine einzelne Base gegen eine andere ausgetauscht war - lediglich 1,44 Prozent beträgt. Daneben fanden wir aber fast 68.000 längere Abschnitte, in denen ganze Basenfolgen als Insertion eingebaut oder als Deletion verloren gegangen sind. Die Aminosäuresequenz der Proteine unterscheidet sich damit in 83 Prozent der 231 auf Chromosom 21 (Mensch) bzw. 22 (Schimpanse) entdeckten Gene. Während sich viele Proteine bei Schimpanse und Mensch nur geringfügig unterscheiden, zeigen sich bei ca. 20% strukturelle Unterschiede, die zu andersartigen Funktionen führen können. Wenn man berücksichtigt, dass die Chromosomen 21 bzw. 22 nur etwa ein Prozent des gesamten Erbguts tragen, könnten sich die Unterschiede zwischen Mensch und Schimpanse auf mehrere tausend Gene erstrecken. Die genetischen Abweichungen zwischen Mensch und seinem nächsten Verwandten wären damit viel größer und komplexer als bisher vermutet. (An diesen Arbeiten war neben unserer Abteilung auch die Abteilung „Evolutionäre Genetik“ (Svante Pääbo) des Max-Planck-Instituts für evolutionäre Anthropologie in Leipzig beteiligt.)
Ein weiterer Fokus unser Arbeiten liegt in der Analyse des menschlichen X-Chromosoms, dessen (weitgehend) endgültige Sequenz in diesem Jahr veröffentlicht werden konnte [7]. Die Arbeiten an der Sequenzierung ausgewählter Regionen des Geschlechtschromosoms X des Schimpansen wurden bereits begonnen. Das X-Chromosom ist wegen seiner großen Anzahl an Genen, die mit Erbkrankheiten in Verbindung gebracht werden, von besonderem medizinischen Interesse, speziell für verschiedene Formen geistiger Retardierung (vergleiche H.-Hilger Ropers, Molekulare Ursachen genetisch bedingter kognitiver Störungen, Jahrbuch der MPG 2004).
Funktionelle Genomanalyse der Gene auf Chromosom 21
Ein Großteil des medizinischen Interesses am Chromosom 21 beruht auf Studien der Trisomie 21 (Down-Syndrom, DS). Unser Ziel ist es, einen Beitrag zum Verständnis der molekularen Basis des DS-Krankheitsbildes zu leisten. Um neue Marker von Trisomie 21 zu identifizieren, haben wir Genexpressionsprofile von menschlichen trisomischen Zellen mittels cDNA-Chips hergestellt, die ca. 16.000 Gene umfassen. Da der Zugang zu menschlichen Proben begrenzt ist, arbeiten wir mit einem etablierten Mausmodell für Trisomie 21 (Ts65Dn), das die Hälfte der dem Chromosom 21 äquivalenten Gene in der Maus in dreifacher Kopie trägt und mehrere der DS-Merkmale ausprägt. Tiermodelle sind essenzielle Werkzeuge, um die Komponenten komplexer Krankheiten zu untersuchen. Wir haben die Genexpressionsprofile von mehreren Geweben dieser trisomischen Mäuse mittels cDNA-Chips und quantitativer PCR ermittelt und konnten so neue Erkenntnisse über das Expressionsverhalten von Genen in einem trisomischen Kontext gewinnen [8]. Um die Funktion der Proteinprodukte der Chromosom 21-Gene systematisch zu untersuchen, wurden bislang zwei Drittel der Gene kloniert und in Bakterien exprimiert. Dies dient u. a. zur Produktion von Antikörpern (Abb. 1).
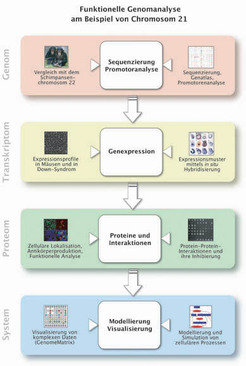 Bild vergrößern
Bild vergrößern
Wir konnten 54 Antikörper gewinnen, darunter viele gegen Proteine mit unbekannter Funktion. Unsere Kollektion an Proteinen und Antikörpern für Chromosom 21 dient darüber hinaus dem Test des Designs und der Erprobung von Anwendungsmöglichkeiten neuer Techniken in der Proteomforschung, wie etwa Protein- und Antikörperchips. Unsere Chromosom 21-Genkollektion haben wir sowohl institutsintern als auch im Rahmen des Nationalen Genomforschungsnetzes (NGFN) einer Reihe von Forschungsgruppen zur Verfügung gestellt, um weitergehende Informationen zu den Genfunktionen zu erhalten. So wurde die zelluläre Lokalisation von 89 der klonierten Genprodukte bestimmt. Für 52 Proteine, von denen ein großer Teil möglicherweise an der Entstehung des Down-Syndroms beteiligt ist, konnte die Lokalisation in HEK293-Zellen bestimmt werden. Über Affinitätsreinigung konnten weitere Proteinkomplexe ermittelt werden, an denen auch Proteine vom Chromosom 21 beteiligt sind.
In Zusammenarbeit mit Erich Wanker (Max-Delbrück Centrum für Molekulare Medizin (MDC), Berlin-Buch) wurde eine Suche nach Interaktionspartnern von Chromosom 21-Proteinen durchgeführt. Der erste automatisierte Yeast-2-Hybrid-Screen mit 71 Proteinen ergab 52 Interaktionen für 22 Proteine. Diese konnten zu einem großen Teil in anderen Versuchen bestätigt werden. Die Integration unserer Expressions-, Lokalisations- und Interaktionsdaten erleichtert nun die Suche nach Kandidatengenen für das Down-Syndrom. Immer mehr Teile fügen sich auf diese Weise langsam zu einem erkennbaren Bild dieser komplexen Krankheit zusammen.
Einen weiteren wichtigen Beitrag erhoffen wir uns von der Anwendung systembiologischer Ansätze. Diese sollen es ermöglichen, die komplexen Prozesse besser zu verstehen, die durch die Änderung der Anzahl an Chromosomen 21 in der Zelle verursacht werden. Möglicherweise leisten sie auch einen Beitrag zur besseren Früherkennung dieses Leidens beziehungsweise zur Entwicklung von Methoden, durch die die Lebenssituation der Betroffenen verbessert werden kann. Down-Syndrom und Chromosom 21 sind auch ein erster Schritt zum besseren molekularen Verständnis vieler anderer, weit häufigerer Erkrankungen wie zum Beispiel Krebs, bei denen wir die an der Krankheit beteiligten Gene noch nicht so eindeutig identifizieren können. Wir hoffen, durch die Kombination von funktioneller Genomforschung und Systembiologie einen Beitrag zur verbesserten Diagnose und Therapie von Krebserkrankungen leisten zu können.