Struktur und Funktion von Membranproteinen
Forschungsbericht (importiert) 2010 - Max-Planck-Institut für Biophysik
Einführung
Membranproteine gehören zu den faszinierendsten Molekülen in lebenden Zellen. In den Lipidmembranen, die jede Zelle umgeben und unterteilen, sind Membranproteine für eine große Anzahl verschiedener biologischer Prozesse zuständig, von denen viele lebenswichtig sind. Ein genaues Verständnis jedes einzelnen dieser Prozesse erfordert die Kenntnis der exakten räumlichen Struktur der beteiligten Proteine, möglichst im atomaren Maßstab. Oft müssen die Strukturen von zwei oder mehr verschiedenen funktionalen Zuständen aufgeklärt werden. Das gilt ganz besonders für Membranproteine, deren Funktion häufig auf Konformationsänderungen beruht.
Das Max-Planck-Institut für Biophysik ist ein weltweit bekanntes Zentrum für die Erforschung von Membranproteinen, insbesondere ihrer Struktur und molekularen Mechanismen. Die Abteilung Strukturbiologie ist besonders an Proteinen und großen Komplexen interessiert, die im Membrantransport, in der Photosynthese oder der Zellatmung aktiv sind. Die Forscher untersuchen die Struktur dieser Proteine mit Elektronen- oder Röntgenkristallographie, sowie mittels Kryo-EM von Einzelpartikeln. Darüber hinaus wird die Struktur, Anordnung und Verteilung dieser Proteine und Komplexe in der Membran mit Hilfe der Elektronen-Kryotomographie untersucht.
Das Spektrum an untersuchten Membranproteinen und Komplexen ist weit. Im Folgenden soll an einigen Beispielen die aktuelle Forschung dargestellt werden.
Strukturen von Antiportern

Seit annähernd zehn Jahren beschäftigt sich die Gruppe mit der Aufklärung der hochaufgelösten Struktur des Substrat-Produkt-Antiporters CaiT. Dieser Antiporter, der über die Zellmembran L-Carnitin gegen g-Butyrobetain tauscht, ist ein Beispiel für viele Ionen-unabhängige Sekundärtransporter in der Zelle: Im Gegensatz zu anderen sekundären Transportern hängt die Aktivität von CaiT nicht von einem Ionen- oder Protonengradienten ab. Nun konnten zwei Strukturen von CaiT gelöst werden: mit einer Auflösung von 2.3 Å die aus dem Enterobakterium Proteus mirabilis (PmCaiT) und mit einer Auflösung von 3.4 Å Auflösung aus E. coli (EcCaiT). EcCaiT weist dabei zwei gebundene Substratmoleküle auf, eines in der zentralen Bindungsstelle, das andere in einer neu entdeckten extrazellulären Bindungsstelle (Abb 1). Beide Bindungsstellen sind in der Struktur von PmCaiT leer. Der Vergleich zeigt, dass in PmCaiT eine Tryptophan-Seitenkette um 60 Grad verdreht ist, was die Substratbindung in der zentralen Bindungsstelle verhindert. Substratbindungs- und Transportuntersuchungen in Proteoliposomen zeigen, dass der Transport kooperativ ist – ein Novum für Sekundärtransporter [1].
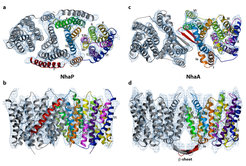
Ein weiteres langfristiges Interesse der Forschungsgruppe gilt der Bestimmung der Struktur und molekularen Funktion von Natrium-Protonen-Antiportern. Unter anderem werden Untersuchungen von Substrat- und pH-induzierten Konformationsänderungen an NhaA aus E. coli [2] und dem verwandten Na+/H+-Antiporter NhaP1 von Methanococcus jannaschii durchgeführt. Jüngst wurde eine 7 Å - 3D-Karte von M. jannaschii NhaP1 erhalten [3], die sowohl wichtige Ähnlichkeiten als auch Unterschiede zu der von NhaA zeigt (Abb. 2).
Transporter gegen Stress
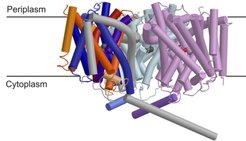
Die Minerva-Forschungsgruppe um Christine Ziegler geht der Frage nach, wie die Stressantwort-Mechanismen in Prokaryonten (einschließlich Pathogenen) und Eukaryonten aussehen. Zellen passen sich generell einer Vielzahl von Stressmechanismen an. So gewährleistet ein komplexes Netzwerk regulatorischer Systeme eine koordinierte und effektive Antwortkaskade, indem Genexpression und Proteinaktivität verändert werden. Es werden hauptsächlich Membrantransporter, die in die Stressantwort involviert sind, untersucht. Diese Transporter können Stress-Stimulantien erkennen, sie in Zell-relevante Signale verwandeln und ihr Aktivitätsprofil verändern. Das am meisten untersuchte Projekt bezieht sich auf den molekularen Mechanismus der Wahrnehmung und Regulierung osmotischer Signale im Na+/Betain-Transporter BetP von Corynebacterium glutamicum. Es konnte die Struktur von BetP mittels Röntgenkristallographie bei einer Auflösung von 3.35 Å gelöst werden (Abb. 3), [4], durch Elektronenkristallographie an 2D-Kristallen wurde eine 3D-Karte mit einer Auflösung von 8 Å erhalten, die zeigt, dass im BetP-Trimer alle drei Protomere verschiedene Konformationen aufweisen, was für die Regulierung des Transports wichtig ist.
Superkomplexe der Atmungskette

Vor einigen Jahren wurde damit begonnen, die Struktur mitochondrialer Superkomplexe der Atmungskette mit Hilfe der cryo-EM zu untersuchen. Diese Arbeit wurde mit der Einzelpartikel-cryo-EM am Superkomplex fortgesetzt, der nach einem neuen Dichtegradienten-Reinigungsprotokoll isoliert wurde und eine höhere Auflösung von 19 Å erzielte (Abb. 4). In diese Karte konnten die Röntgenstrukturen des Rinder-Komplex-III-Dimers, des Rinderherz-Komplex-IV-Monomers und des kürzlich veröffentlichten Komplexes I von Yarrowia lipolytica eingepasst werden. Die Anordnung der Atmungskettenkomplexe im Superkomplex ist optimal für effizienten Elektronentransport, sowohl durch Ubiquinol zwischen den Komplexen I und III, als auch durch Cytochrom c zwischen den Komplexen III und IV.
ATP-Synthase bildet Dimer-Reihen

Schon die ersten Elektronen-kryotomographischen Studien an mitochondrialen Membranen vor vier Jahren zeigten, dass die F1-Fo-ATP-Synthase aus Rattenleber- und Rinderherz-Mitochondrien unerwarteterweise nicht zufällig verteilt vorlag, sondern dass sie lange Dimer-Reihen in der Membran bildete [5]. Diese Dimer-Reihen waren immer mit Regionen hoher Membrankrümmung verknüpft. Seitdem wurden die Studien ausgeweitet und andere Spezies sowie ganze Mitochondrien untersucht. So wurden z.B. mit Cryo-ET Dimer-Reihen in intakten Organellen vom Rinderherz und jungen Podospora anserina-Zellen gefunden (Abb. 5).
Bei der Untersuchung von Mitochondrien oder isolierten Cristae-Vesikeln von insgesamt sechs verschiedenen Spezies (2 Säuger, 2 Hefen, 1 filamentöser Pilz, 1 Pflanze) fand man heraus, dass die Dimer-Reihen der ATP-Synthase eine universelle und fundamentale Struktur in allen eukaryontischen Zellen darstellen. Die funktionelle Bedeutung dieser Dimer-Reihen liegt darin, dass sie kinetische Protonenfallen bilden, die eine effiziente ATP-Synthese gewährleisten.
ATP-Synthase der Archaebakterien
Die Projektgruppe von Janet Vonck befasst sich unter anderem mit der A-Typ-ATP-Synthase von archaea, die sich von F1F0-Komplexen in Bakterien, Mitochondrien und Chloroplasten unterscheidet. Sie ist eher mit den V-Typ-ATPasen verwandt und zeigt wie diese multiple periphäre Statorproteine (stalks). In Zusammenarbeit mit Volker Müller von der Universität Frankfurt am Main wurde die A-Typ-ATP-Synthase aus dem hyperthermophilen Pyrococcus furiosus mit Einzelpartikel-EM untersucht. Die Rekonstruktion bei einer Auflösung von etwa 23 Å zeigte klar die Präsenz zweier periphärer Statorproteine. Der K-Ring, bestehend aus vier Helix-Untereinheiten mit nur einer Ionenbindungsseite, wurde klar sichtbar. Die massenspektrometrische Analyse ergab ein Molekulargewicht von 730 kDa, was mit einer Stöchiometrie von A3B3CDE2FH2ac10 übereinstimmt. Die Röntgenstrukturen von A-Typ-ATPase-Untereinheit wurden in die EM-Karte eingepasst, so dass ein nahezu komplettes Modell des Komplexes entstand [6] .
Die Rotorringe der ATP-Synthase
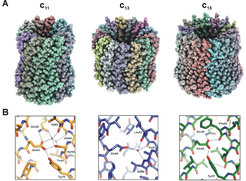
Auch in der selbständigen Forschungsgruppe um Thomas Meier steht im Mittelpunkt die ATP-Synthase, der Hauptlieferant der universellen Energie in Form von Adenosintriphosphat (ATP) in den Zellen. Dieses Enzym arbeitet als molekularer Motor und besteht aus einem Stator und einem Rotor. Seit 2006 arbeitet die Gruppe an der strukturellen und funktionellen Charakterisierung der Rotor-Ringe im Fo-Komplex (Abb. 6).
Kürzlich gelang es, die Struktur des ersten Protonen-bindenden c15 Rotor-Rings mit hoher Auflösung aufzuklären, welcher aus dem Cyanobakterium Spirulina platensis isoliert wurde [7]. Außerdem entdeckte die Gruppe einen neuen Typ von Protonenbindung in ATP-Synthasen [8]. So gewannen die Forscher beispiellose Einblicke in den Mechanismus, mit dem die Ionen an die ATP-Synthase-Rotoren binden und der darauffolgende Ionenaustausch stattfindet. Außerdem konnte man den Mechanismus aufklären, der der Ionentranslation und dem Prozess des entstehenden Drehmoments in der Fo ATP-Synthase unterliegt – auf Basis von Kristallstrukturen, deren Auflösung unterhalb von 2 Å liegt [9].
![Struktur von EcCaiT mit zwei gebundenen Substratmolekülen. Ein Querschnitt durch das Protomer zeigt den offenen cytoplasmatischen Trichter und die gebundenen g-Butyrobetainmoleküle (Pfeile) [1]. Struktur von EcCaiT mit zwei gebundenen Substratmolekülen. Ein Querschnitt durch das Protomer zeigt den offenen cytoplasmatischen Trichter und die g](https://www.mpg.de/1245157/original-1302008399.jpg?t=eyJ3aWR0aCI6MzQxLCJmaWxlX2V4dGVuc2lvbiI6ImpwZyIsIm9ial9pZCI6MTI0NTE1N30%3D--4f78aa04d662f110c3ef02ed35edacd066e1b3a4)
![Modell der Struktur von NhaP1, das der 7 Å-Elektronenkristallographischen Karte angepasst wurde und auf dem Homologieabgleich mit der NhaA-Sequenz und auf Röntgenstruktur-Kristalldaten basiert [3]. Zusätzliche Helix 1 in rot. a) Ansicht vom Cytoplasma; b) Seitenansicht. Modell der Struktur von NhaP1, das der 7 Å-Elektronenkristallographischen Karte angepasst wurde und auf dem Homologieabgleich mit der NhaA-Sequenz u](https://www.mpg.de/1245311/original-1302008406.jpg?t=eyJ3aWR0aCI6MzQxLCJmaWxlX2V4dGVuc2lvbiI6ImpwZyIsIm9ial9pZCI6MTI0NTMxMX0%3D--b4ab40c41b1b40983129b75a36d1f6c5c6ed91f7)
![Seitenansicht des BetP-Trimers. Die Wahrnehmung osmotischer Signale der C-terminalen Domänen sind zur cytoplasmatischen Seite des jeweils benachbarten Protomers orientiert. Dabei bildet sich ein interaktives Netzwerk zwischen den Aminosäureresten der cytoplasmatischen Schleifen 2 und 8 mit der C-terminalen Domäne des benachbarten Protomers [4]. Seitenansicht des BetP-Trimers. Die Wahrnehmung osmotischer Signale der C-terminalen Domänen sind zur cytoplasmatischen Seite des jeweils benachbart](https://www.mpg.de/1245448/zoom-1302008414.jpg?t=eyJ3aWR0aCI6MzQxLCJmaWxlX2V4dGVuc2lvbiI6ImpwZyIsIm9ial9pZCI6MTI0NTQ0OH0%3D--b7a7051433efea4a15692010b528f49c772ba45b)


![Strukturen der Na- und Wasserstoffionen-bindenden Rotor-Ringe von ATP-Synthasen: Der Na+ bindende c11 Rotor-Ring von Ilyobacter tartaricus, der H+ (und H2O) bindende Bacillus pseudofirmus OF4 c13 Rotor-Ring [8] und der H+ bindende Spirulina platensis c15 Rotor-Ring [7]. (A) Gesamtansicht mit der cytoplasmatischen Seite oben; (B) entsprechende Ionenbindungsstellen. Strukturen der Na- und Wasserstoffionen-bindenden Rotor-Ringe von ATP-Synthasen: Der Na+ bindende c11 Rotor-Ring von Ilyobacter tartaricus, der H+ (un](https://www.mpg.de/1343856/original-1302008589.jpg?t=eyJ3aWR0aCI6MzQxLCJmaWxlX2V4dGVuc2lvbiI6ImpwZyIsIm9ial9pZCI6MTM0Mzg1Nn0%3D--d8586c6deaea4a1c7ae8a83eafab13eb0d7cff00)