
Software der Abteilung für Theoretische Biophysik
BioEM
Bayesian Inference Of Electron Microscopy
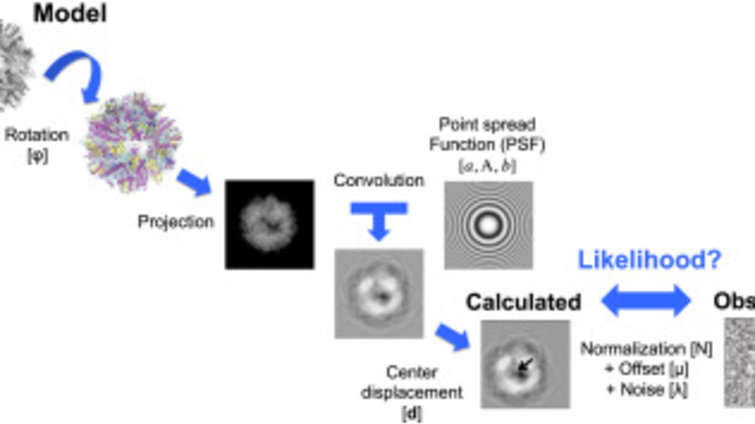
Beschreibung: Die Methode ermöglicht die Charakterisierung von Strukturmodellen durch Berechnung ihrer posterioren Wahrscheinlichkeit in Bezug auf einzelne EM-Bilder.
Beschreibung: Die Methode ermöglicht die Charakterisierung von Strukturmodellen durch Berechnung ihrer posterioren Wahrscheinlichkeit in Bezug auf einzelne EM-Bilder.





